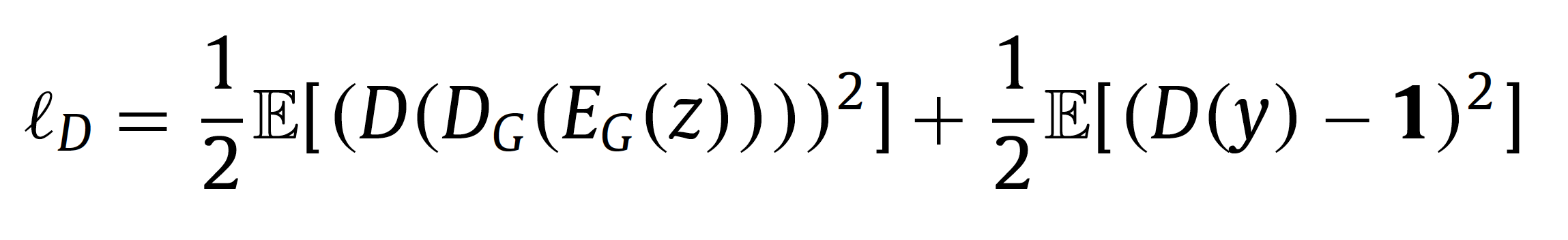[Paper Review] A disentangled generative model for disease decomposition in chest X-rays via normal image synthesis_1편
![[Paper Review] A disentangled generative model for disease decomposition in chest X-rays via normal image synthesis_1편](/content/images/size/w2000/2020/11/Title.png)
-
Anomaly Detection에 어느정도 관련된 논문이며 DGM으로 abnormal CXR이 입력으로 들어오면 정상 이미지와 비정상 영역을 분리해서 질병,질환이 있는 부분을 검출하고 시각화 할 수 있는 방법을 보여주는 논문입니다.
-
2020년도 Medical Image Analysis에 publish된 논문이며 Impact Factor는 11.280입니다.
-
Paper 원문: A disentangled generative model for disease decomposition in chest X-rays via normal image synthesis
-
Anomaly Detection에 관련된 논문인 F-AnoGAN과 AnoGAN와 함께 비교해가며 읽어 보시는것을 추천드립니다.
Abstract
[medical images의 해석이 어려운 이유]
medical images의 해석은 cautious observation, 정상적인 신체 해부학에 대한 정확한 이해 및 분석, 생리학 및 병리학의 지식들을 요구하는 복잡한 combining knowledge이다. Chest X-rays(CXR) 영상을 해석하는 것은 2D-CXR 영상이 해상도가 낮고 경계가 불분명한 organs/tissues 사이에 대한 중첩을 보여주기 때문에 medical images의 해석이 어렵다.
[Deep Generative Model(DGM)을 제안]
질병 진단(diagnosis) / 분류(classification)에 초점을 맞춘 과거의 CXR 컴퓨터 보조 진단 작업과 다르게 본 논문에서는 먼저 입력된 abnormal CXR 이미지에서 abnormal disease residue maps과 normal CXR 이미지를 동시에 생성하는 Deep Generative Model을 제안한다.
[DGM의 직관적인 설명]
제안한 방법의 직관은 질병 영역이 일반적으로 abnormal CXR에서 normal tissues의 pixel 위에 겹쳐지거나 대체된다는 가정을 기반으로 한다. 따라서 질병 부위는 생성된 환자 특유의 normal CXR과 비교함으로써 abnormal CXR에서 disentangled 또는 decomposed될 수 있다.
[DGM 구성]
Deep Generative Model(DGM)은 3개의 Encoder-Decoder로 구성 되어있다.
- Adversarial Learning을 사용한
normal CXR 이미지 합성을 위한 것. - disease separation을 위한
residue maps을 생성하여 abnormal region을 묘사. - 훈련 과정을 촉진하고 noisy data에 대한
모델의 견고성을 향상시키기 위한 것이다.
[reconstruction loss]
self-reconstruction loss는 두 지점에서 생성된 normal CXR 이미지를 적용하여 original CXR과 시각적으로 유사한 구조를 갖도록 역할을 하는 loss다.
[평가 방법]
우리는 large scale CXR dataset을 통해 우리의 모델을 평가했다.
[결과 & 응용 방법]
결과는 우리의 모델이 사실적이고 normal CXR 영상과 함께 질병 residue/saliency map을 생성할 수 있다는 것을 보여준다. disease residue/saliency map은 방사선사가 임상 실습에서 CXR 판독 효율을 개성하기 위해 사용할 수 있다. synthesized normal CXR은 맞춤형 경도 질환 연구(personalized longitudinal disease study)의 data augmentation 및 normal control에 사용될 수 있다.
또한 DGM은 normal/abnormal CXR classifiaction, Lung opacity classification/detection을 포함한 몇 가지 중요한 임상적 용도에 대해 진단 성능을 정량적으로 향상 시킨다.
Introduction
- Chest X-ray 검사는 병원에서 가장 일반적으로 이루어지는 검사이며 CT,MRI보다 방사선 노출이 가장 적고 스캔 시간이 적으며 비용도 가장 저렴한 검사 방법입니다. 검사 시간은 다음과 같습니다.
Chest X-ray < 15분, CT < 15 ~ 30분, MRI 10 ~ 60분
- 초기에는 전문가들의 작업 부하 감소를 위해 CXR에 대한 컴퓨터 지원 진단/검출 방법인 Developing computer-aided diagnosis/detection(CADx/ CADe) 방법을 개발하고 사용했으며 많은 이득을 얻었다고 합니다.
- 최근 몇 년간은 CXR image data & NLP을 사용한 text-mined labels이 제공되어 Neural Network을 이용하는 제품들이 많이 제안되 었습니다. 예를 들어보면 아래와 같습니다.
위에 언급된 예시들의 논문을 보면 2020년도 최근에 나온 논문도 있다는 것을 확인할 수 있습니다. 이런 방법돌의 결과도 괜찮았지만 CXR 영상에 대한 딥러닝의 해석 가능성을 개선하기 위한 연구가 매우 제한적이라는 단점이 존재했습니다.
위 주장에 대한 논문 입니다 ... https://journals.plos.org/plosmedicine/article?id=10.1371/journal.pmed.1002707
따라서 이런 문제점들을 해결하기 위해 본 논문에서 제안한 DGM(Deep Generative Model)에 대한 내용을 아래에서 설명하도록 하겠습니다.
Deep disentangled generative model(DGM)
DGM의 역할
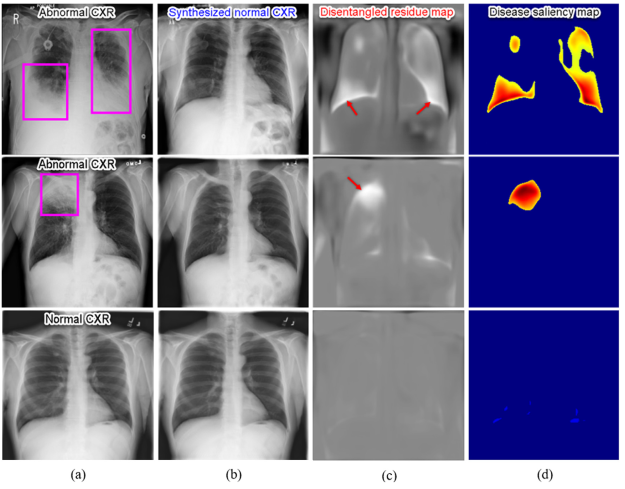
DGM은 abnormal CXR이 query data로 입력으로 들어오면 synthesized normal CXR(b) 및 disentangled residue map(c)으로 decompose하기 위해 disentangled Representation Learning Framework을 개발했습니다.
대부분의 Unsupervised 로 이루어지는 Anomaly Detection에서 공통적으로 접근하는 방식으로 query data로 비정상이 들어오면 그것을 정상 이미지(Synthesized normal CXR)로 생성을 해줘야 합니다.
특이한 점은
3개의 Encoder-Decoder로 구성된 DGM(Deep Generative Model)이 (b)와 (c)로 decompose해서 질병 영역과 정상 영역으로 분해한다는 특징이 있습니다.
abnormal CXR입력으로 들어오면 network는 (그림 1)의(b)와 같이 decompose된 Synthesized normal CXR로 생성을 해줘야 합니다. 즉, query image로 어떤 영상이 들어오든 network는 정상 이미지를 생성해야 합니다.
하지만...
아쉽게도 이 논문에서는 정확하게 어떤 Network로 Decompose된 Synthesized normal CXR를 생성 해주었는지 언급은 되어있지 않습니다.
Deep disentangled generative model에 의해 얻어진 Disentangled residue map(c)을 normalized하고 disease saliency map 또는 attention map(d)을 효과적으로 시각화 시킬 수 있으므로 방사선사 및 전문의가 이상 탐지에 쉽게 집중할 수 있도록 도와줄 수 있게 됩니다.
(그림 1)에 대해 자세하게 한번 설명을 해보도록 하겠습니다.
(그림 1)은 CXR synthese 및 dianosis을 위해 제안된 disentangled generative deep model의 예시를 시각적으로 보여주는 그림 입니다. (a)abnormal/normal CXR 이미지가 입력 되면 (c)잠재적 질병 패턴(potential disease patterns)을 얻기 위해 DGM이 입력된 CXR을 (b)synthese normal CXR와 (c)residue map으로 disentangling 하게 됩니다.
(c)disentangled residue maps을 normalized하고 (d)disease saliency/attention map을 효과적으로 시각화 할 수 있으므로 방사선사가 이상 영역에 쉽게 집중할 수 있도록 도와줄 수 있게 됩니다.
Saliency map이란?
pixel의 밝기 또는 두드러지는 정도를 나타에는 map으로써 사람이 볼 때 밝은 부분이 더 강조되는 것처럼 sliency map도 시각적으로 강조가 되는 부분입니다. Heatmap이라고 이해하시면 좋을거 같습니다.
(참고 링크: https://www.geeksforgeeks.org/what-is-saliency-map/)
본 논문에서 제안한 방법으로 어떻게 sliency map을 생성하는지 알아보도록 하겠습니다.
saliency map 생성 방법
- Lung segmentation 부위로 부터 bounding box(a의 1,2행) 을 정의
- adaptive threshold을 사용해서 residue map을 이진화.
- saliency map을 시각화 하기 위해 bounding box(a의 1,2행)와 overlapped된 connectioned components만 표시
정리를 해보자면...
- query image로 abnormal/normal이 들어오면 무조건 normal로 생성해주는 Network를 사용합니다.(이거는 어떤 네트워크를 사용했는지 언급이 안되어 있습니다.)
- DGM은 normal CXR로 생성해주는 synthesized normal CXR & disentangled residue map으로 decompose 해줍니다.
- residue map을 normalize 해서 saliency map을 구해서 시각화를 진행합니다.
CXR decomposition using a desentangled generative model
학습 목표
-
latent space mapping을 학습 하여 normal CXR을 synthesize하기
- \( G:\) {\( \mathbb{X} \cup \mathbb{Y}\)} \( \rightarrow \mathbb{Y} \)의 출력 값인 Synthesized normal CXR \( y^{'} = G(z)\)가 \( y ∈ \mathbb{Y}\)와 구별 않도록(=완전 똑같이 생기도록) 생성하기. ( \( \mathbb{X}\): abnormal / \( \mathbb{Y}\): normal )
-
\( a = F(z), z^{'} = G(z) + F(z), z^{'} = z \)을 충족하는 mapping \( F\)을 학습하여 residue map \( a\)을 disentangle 하기.
- residue map은 질병을 시각화하는 saliency map, attention map으로 normalize할 수 있다.
비정상 데이터의 정상 데이터를 얻는 방법
input CXR z을 본 논문에서 개발한 disentangled Representation Learning Framework을 이용하여 latent feature space \( L ∈ \mathbb{R}^{n \times k \times k}\)로 인코딩 합니다. 이때 \( n, k\)는 \( L\)의 feature dimenstion입니다.
- latent feature space \( L\)에서의 정상 CXR: \( c_z\)
- \( L\)에서 질병에 대응되는 feature representations: \( c_s\)
Adverarial Learning을 사용해서 GAN을 학습합니다.
\( c_z\)에서 정상적인 CXR \( y\)을 생성하고 \( (y^{'} + a)\)와 \( z\)의 차이가 작아야 한다는 제약 조건을 갖는 \( c_s\)에서 disentangled residue map \( a\)을 생성합니다.
Network Structure
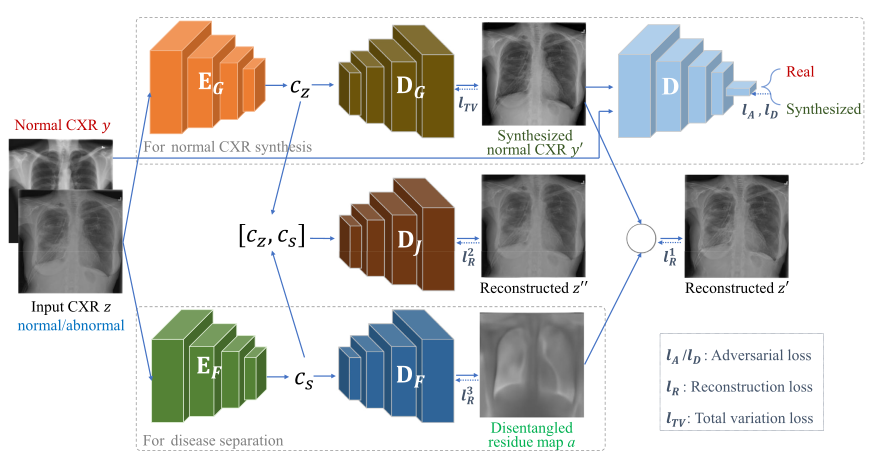
(그림 2)와 같이 제안된 네트워크는 어떻게 보면 \( c_z, c_s\) ∈ L로 정의할 수 있는 \( c_z, c_s\)에서 각각 정상 이미지 \( D_G(c_z)\)와 residue map \( D_F(c_s)\)을 생성할 수 있도록 하기 위해 mapping network \( G, F\)을 학습하기 위한 Framework이며 각 구성 요소들에 대한 정의는 다음과 같습니다.
- \( E_G\): input CXR \( z \rightarrow\) normal CXR 속성을 나타내는 latent feature space \( L\)에서 정상 CXR을 나타내는 \( c_z\)로 인코딩 합니다.
- \( E_F\): input CXR \( z\)을 abnormal을 나타내는 residue map을 질병에 대응되는 latent feature space \( c_s\)로 인코딩 합니다.
- \( D_G\): \( c_z\)에서 \( z\)의 정상 부분을 디코딩 합니다. 이것은 (그림 2)에서 Synthesized normal CXR \( y^{'}\)을 의미합니다.
- \( D_F\): \( c_s\)에서 \( z\)의 비정상 부분을 디코딩 합니다. 이것은 (그림 2)에서 Disentangled residue map \( a\)을 의미합니다.
- \( D_J\): input CXR \( z\)을 \( c_z\)와 \( c_s\)에서 함께 디코딩을 해서 Reconstructed \( z^{''}\)을 의미합니다.
- \( D\): real normal CXR와 \( D_G\)에 의해 생성된 synthesized CXR을 구별하여 True/False로 판단을 하는 역할 입니다.
6개의 요소들 모두 end-to-end 방식으로 학습이 됩니다. 또한 모델 구조를 보명 Encoder와 Decoder는 서로 구조가 유사하지만 weight share는 이루어지지 않고 독립적으로 학습이 진행 됩니다.
Encoder
\( E_G\)와 \( E_F\)는 input CXR z을 dowensampling 하기 위해 여러개의 stride convolutional layers 및 future processing을 위한 여러개의 residal blocks가 포함되어 있습니다.
-
\( E_G\)
모든 Convolutional layers 다음에 Instance Normalization을 사용합니다.그 이유는 Instance Normalization은 중요한 질병 정보를 나타내는 원래의 특징 평균과 분산을 제거해서 정상 이미지로 분리할 수 있게 됩니다.
-
\( E_F\)
- \( E_G\)와는 다르게 질병 영역만 분리해서 encoding을 해줘야 하니까 질병 정보를 제거하는
Instance Normalization을 사용 안합니다.
- \( E_G\)와는 다르게 질병 영역만 분리해서 encoding을 해줘야 하니까 질병 정보를 제거하는
Decoder
\( D_G\)와 \( D_F\)및 \( D_J\)는 latent feature representations \( c_z\), \( c_s\)을 여러개의 residual blocks으로 디코딩 하여 생성된 영상(normal CXR \(y^{'} = D_G(c_z)\)) 및 residue map \( {a} = D_{F}(c_{s})\) 및 reconstructed \( z^{''} = D_J(c_z, c_s)\) 을 생성합니다.
-
\( D_J\)
- MUNIT와 유사하게 MLP을 사용하여 \( c_s\)에서 AdaIN parameter set을 생성하고 생성된 parameter을 기반으로 channel dimenstion에서 \( c_z\)와 \( c_s\)의 concatenation이 AdaIN와 residual blocks에 의해 처리됩니다.
-
\( D\)
- Normal CXR \( y\)와 \( D_G\)가 생성한 Synthesized normal CXR \( y^{'}\)을 구별하는 것을 목표로 합니다.
-
\( D_G\)
- Normal CXR \( y^{'}\)을 생성하려고 하며 multi-scale discriminator을 사용해서 \( D_G\)를 realistic detail과 correct global structure을 보존합니다.
Loss functions
Adversarial loss
진짜같은 가짜 normal CXR을 생성하기 위해 least squares loss을 adversarial learning loss로 사용해서 학습을 진행했습니다.
$$ L_A = \frac{1}{2}\mathbb{E}[(D(D_G(E_G(z))) - 1)^2] $$
Reconstruction loss
DGM은 input CXR \( z\)에서 reconstruction image \( z^{'}\)와 \( z^{''}\)을 생성합니다.
-
\( z^{'} = D_G(E_G(z)) + D_F(E_F(z))\)
-
\( z^{''} = D_J(E_G(z), E_F(z))\)
이때 사용되는 reconstruction loss는 보다 더 선명하게 CXR을 생성하도록 도움을 주는 loss인 L1 distance을 사용해서 각각 아래 수식처럼 정의가 됩니다.
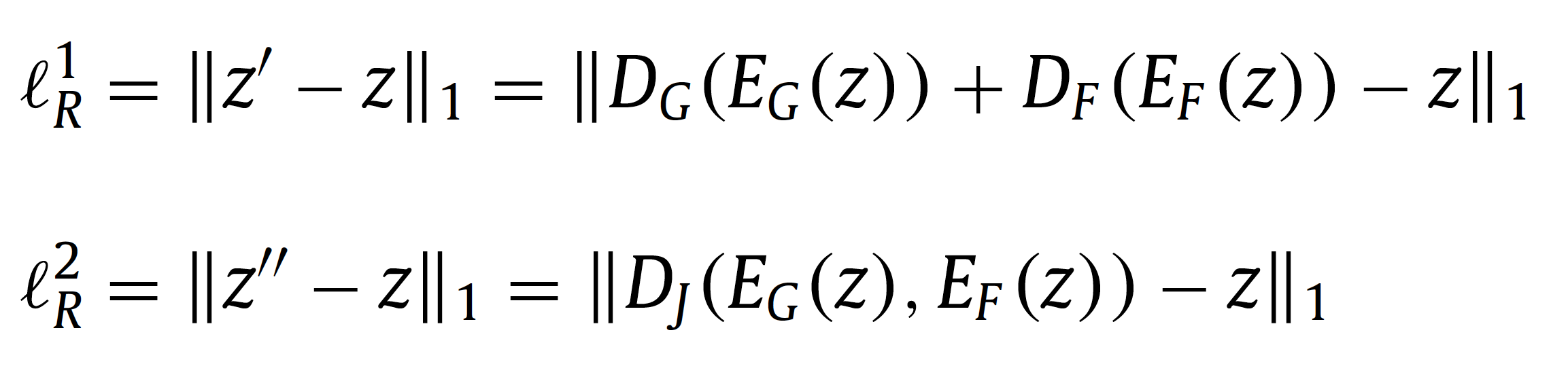
DGM에 input CXR이 정상일 때 loss값이 0인 residue map \( a\)을 생성하게 됩니다.따라서 수식은 다음과 같이 정의됩니다.
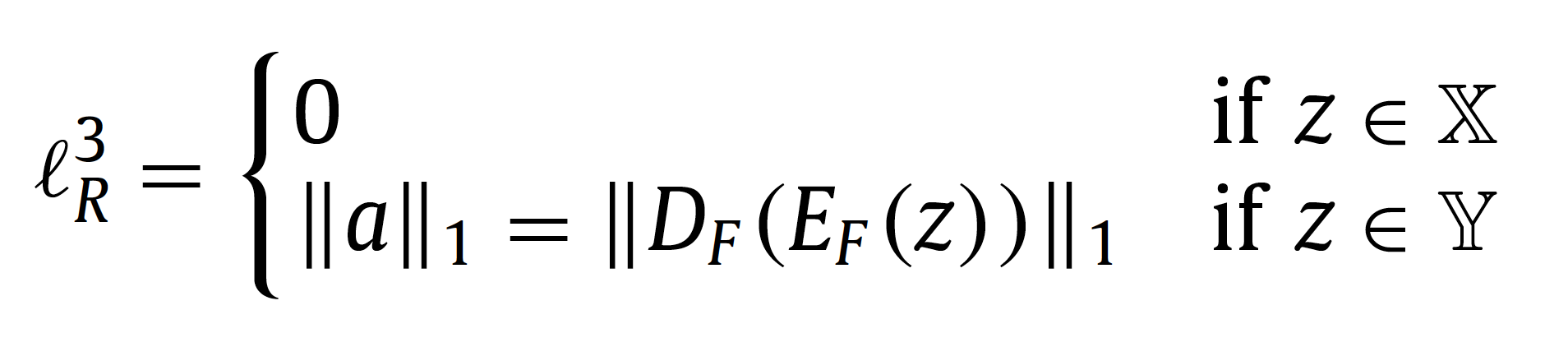
Total variation loss
생성된 정상 CXR인 Synthesized normal CXR \( y^{'}\)[=\( D_G(E_G(z))\)]가 생성될 때 spike artifacts을 줄이고 보다 더 선명하게 생성될 수 있도록 auxiliary loss function인 total varation loss \\( L_TV\\)을 사용하게 됩니다. 수식은 다음과 같이 정의됩니다.
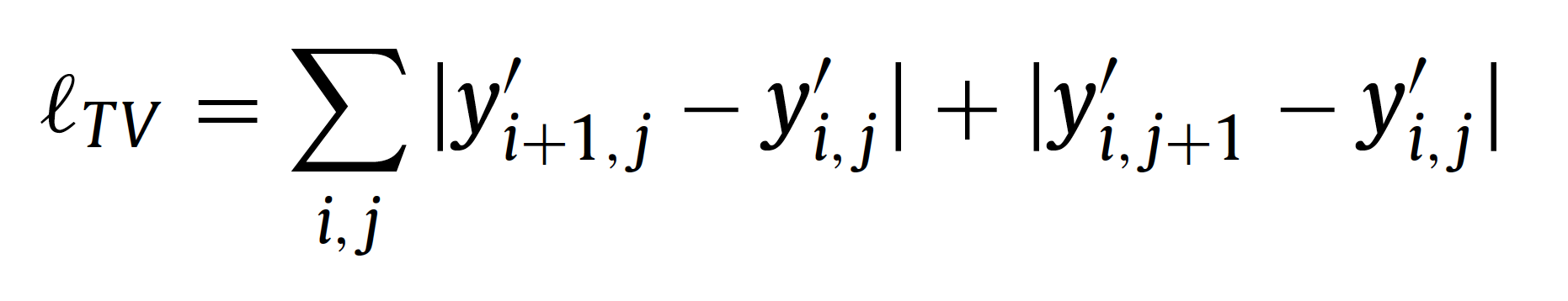
Total encoder-decoder optimization
Encoder-Decoder 구조로 구성된 (\( E_G, D_G, D_J, E_F, D_F)\)들의 optimization의 최종 목표인 \( L(G, F, D_J)\)는 각 Encoder-Decoder에서 나온 loss들의 weighted sum으로 최적화가 진행됩니다.. 수식은 다음과 같이 정의됩니다.
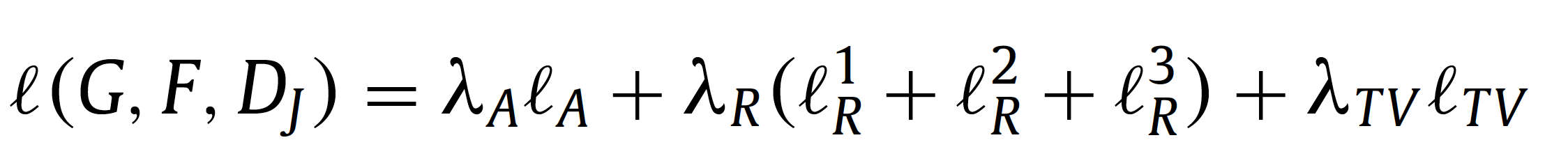
위 수식에서 \( \lambda_{A}\), \( \lambda_{R}\), \( \lambda_{TV}\)는 different losses의 중요성을 control하는 weight입니다.
Discriminator \( D\) optimization
Discriminator \( D\)의 optimization의 경우 LSGAN의 loss function이 사용되었고 그 수식은 다음과 같이 정의됩니다.