[Paper Review] A disentangled generative model for disease decomposition in chest X-rays via normal image synthesis_2편
![[Paper Review] A disentangled generative model for disease decomposition in chest X-rays via normal image synthesis_2편](/content/images/size/w2000/2020/11/title-1.png)
- Anomaly Detection에 어느정도 관련된 논문이며 DGM으로 abnormal CXR이 입력으로 들어오면 정상 이미지와 비정상 영역을 분리해서 질병,질환이 있는 부분을 검출하고 시각화 할 수 있는 방법을 보여주는 논문입니다.
- 1편에 이어지는 내용입니다. 따라서 1편을 먼저 읽고 오시는것을 추천드립니다. 1편 보러가기
Experiments
Dataset
본 논문에서 사용한 데이터는 RSNA Kaggle pneumonia detection challenge에서 사용된 NIH Clinical Center Chest X-ray dataset을 사용했습니다.
train & validation dataset은 25,864개의 frontal view Chest x-ray image를 사용했으며 데이터들의 3가지 타입으로 다음과 같이 구성되어 있습니다.
- normal: 8851개
- lung opacity: 6012개
- 폐렴 및 합병증: 11,821개
테스트 데이터에는 총 1,000개의 frontal view Chest x-ray가 사용 되었습니다.
평가 방법
- AUC score
- sensivity
- specificity
- precision
- F1-score
- average precision
기존 방법과 비교
본 논문에서 제안하는 DGM(Deep Generative Model)은 abnormal CXR을 normal CXR과 residue map으로 decompose 시켜주는 모델 이며 이런 작업은 아직까지 어느 모델도 없다(?)라고 주장을 하고 있습니다.
비슷한 방법으로 image-to-image translation methods을 사용해서 abnormal CXR이 들어오면 그것을 normal CXR로 translation 시켜주는 방법들을 생각해 볼 수 있으며 대표적인 모델로 CycleGAN, MUNIT 및 DRIT이 있습니다.
이 image-to-image translation 모델들은 이용한 abnormal detection은 다음과 같은 방법으로 진행을 할 수 있습니다.
- abnormal CXR이 들어오면 normal CXR로 변형하도록 normal CXR 데이터를 얻는다.
- translation된 normal CXR과 query image(abnormal or normal)와의 차이를 계산해서 residue map을 구한다.
비정상 데이터 분류에 대한 DGM을 평가하기 위해 CNN classification ㅡmodel도 동일한 데이터로 사용함.
Visual ablation study
\( D_J\)와 total varation loss인 \( L_{TV}\)의 효과를 검증하기 위해 2가지 실험을 진행했습니다.
- total varation loss \( L^{2}_{R}\)없이 학습 진행.
- \( L_{TV}\)없이 DGM(Deep Generative Model) 학습 진행.
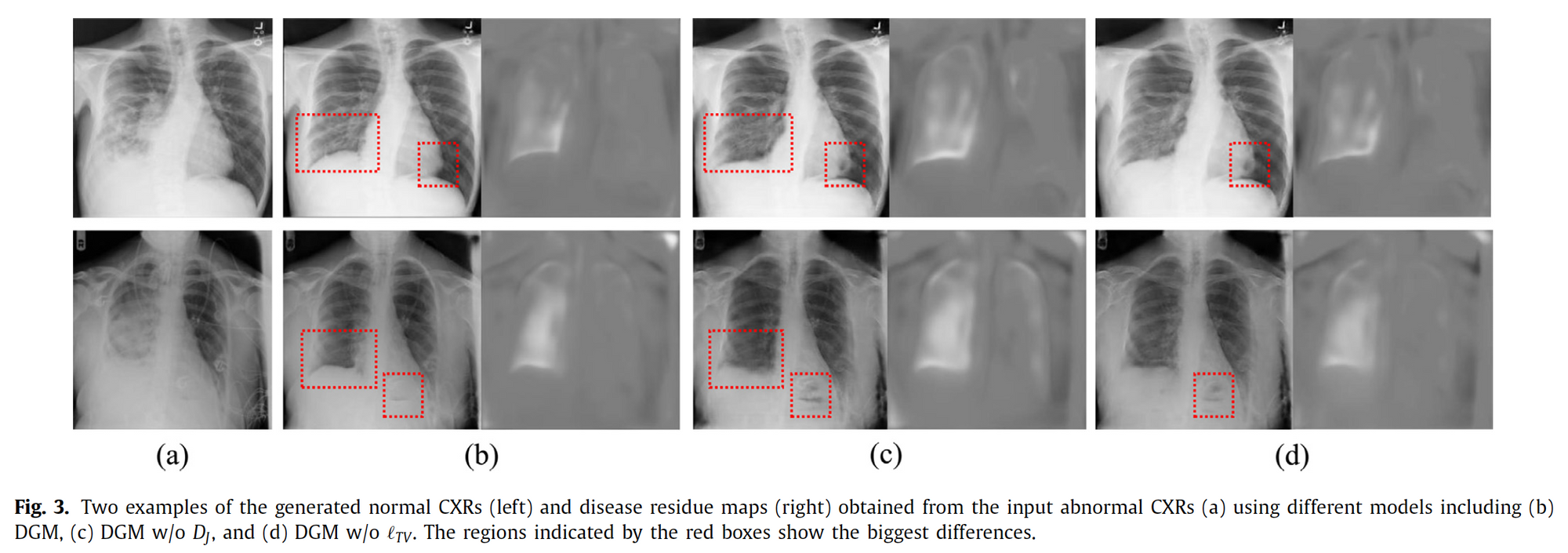
\( D_J\)가 없는 DGM과 비교
\( D_J\)가 있는 DGM이 input CXR data와 구조적으로 유사하게 생성하는 것을 확인할 수 있습니다.
그로 인해 생성된 CXR이 뼈 위치, 쇄골 등과 같이 구조적으로 더 일치하게 생성되어 좀 더 확실한 abnormal regision인 residue map을 만들 수 있습니다.
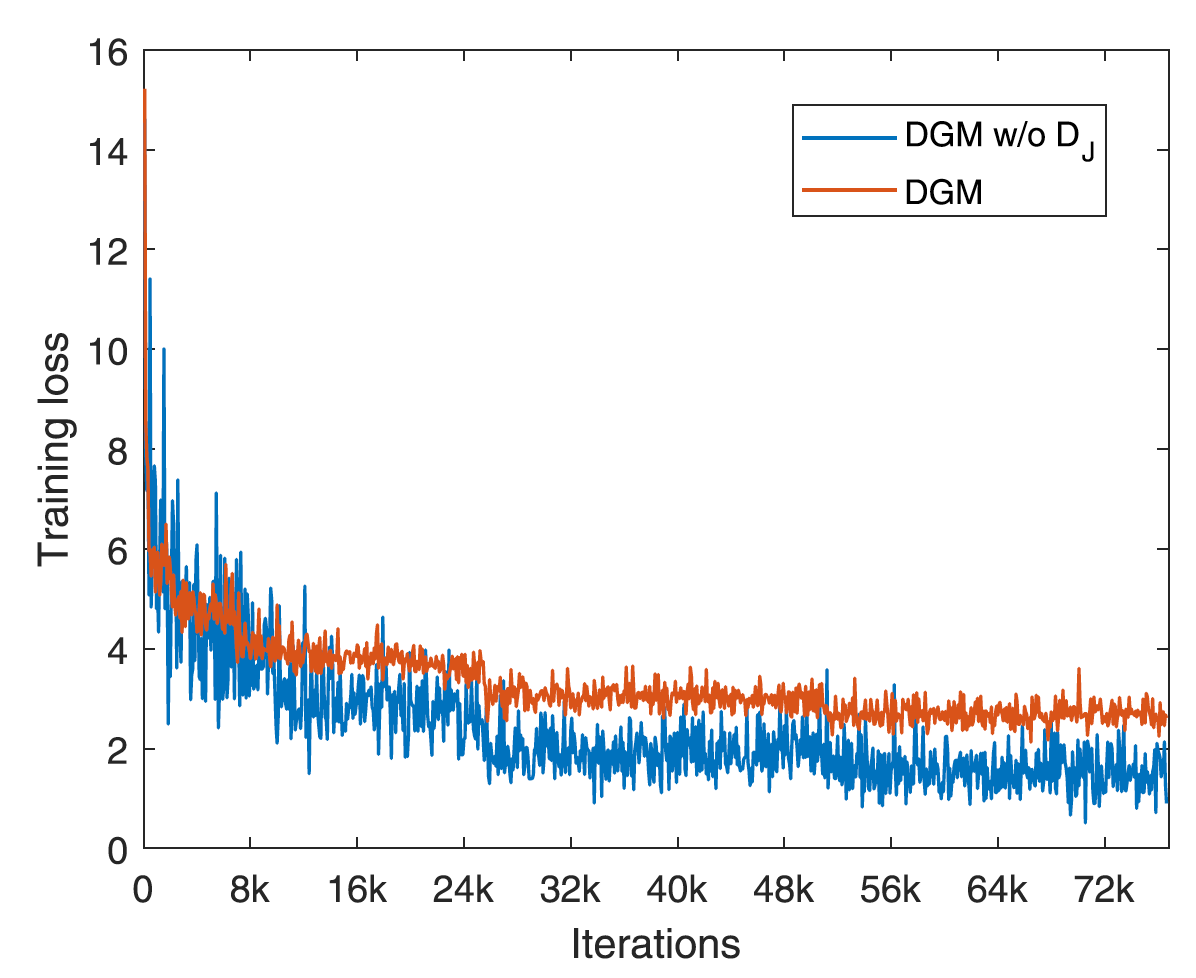
(그림 4)는 train loss을 보여주는 그림인데 \( D_J\)가 있는 붉은색 그래프가 없은 푸른색 그래프 보다 더 안정적으로 빠르게 수렴하는 것을 확인할 수 있습니다.
\( L_{TV}\)가 없는 DGM과 비교
\( L_{TV}\)을 사용하는 DGM이 더 나은 품질과 비교적 적은 artifacts로 normal CXR을 생성하는 것을 확인할 수 있습니다.
결론적으론 \( D_J\)와 \( L_{TV}\)을 사용해야 정확하고 고품질의 영상을 얻을 수 있습니다.
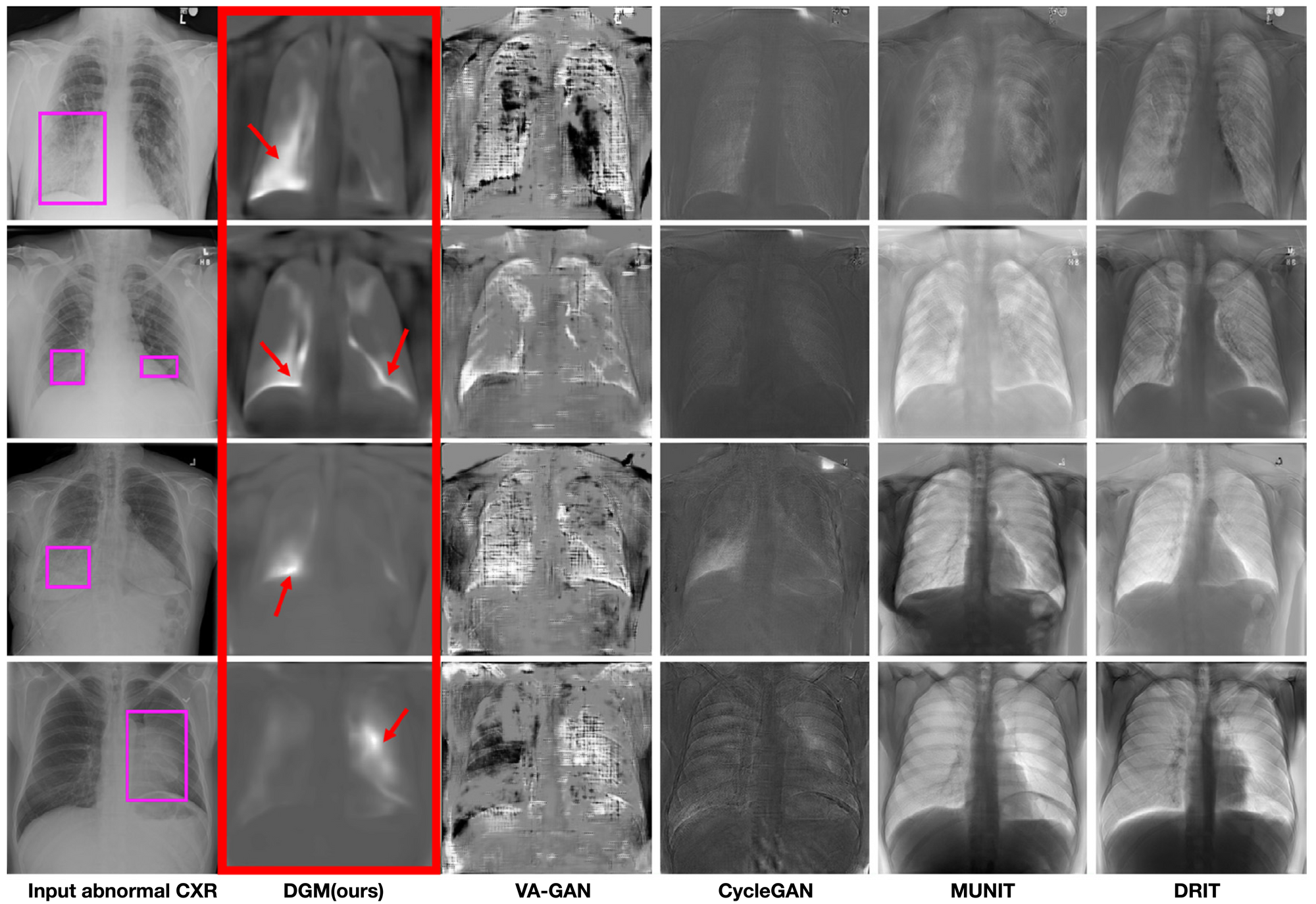
Disentangled disease residue maps vs generated normal CXRs
본 논문에서 제안한 Deep Generative Model이 포함된 disentangled Representation Learning Framework가 얼마나 좋은 성능을 내는지 (그림 5),(그림 6)에 설명을 해두었습니다.
(그림 5)는 Input abnormal CXR이 각 모델을 통해 얻어진 disentangled disease residue map들을 보여주고 있습니다. 당연히(?) DGM이 좋은 결과를 내는 것을 볼 수 있습니다. 또한 본 논문에서 DGM을 통해 disentangled residue map을 이용해서 얻을 수 있는 장점을 다음과 같이 주장하고 있습니다.
- 입력 의료 영상에 대해서 질병에 대한 해석이 가능하다.
- 다른 모델들 보다 더 의미기 있다.
- abnormal regions과 ground truth와 비교를 해보면 가장 일치한다.
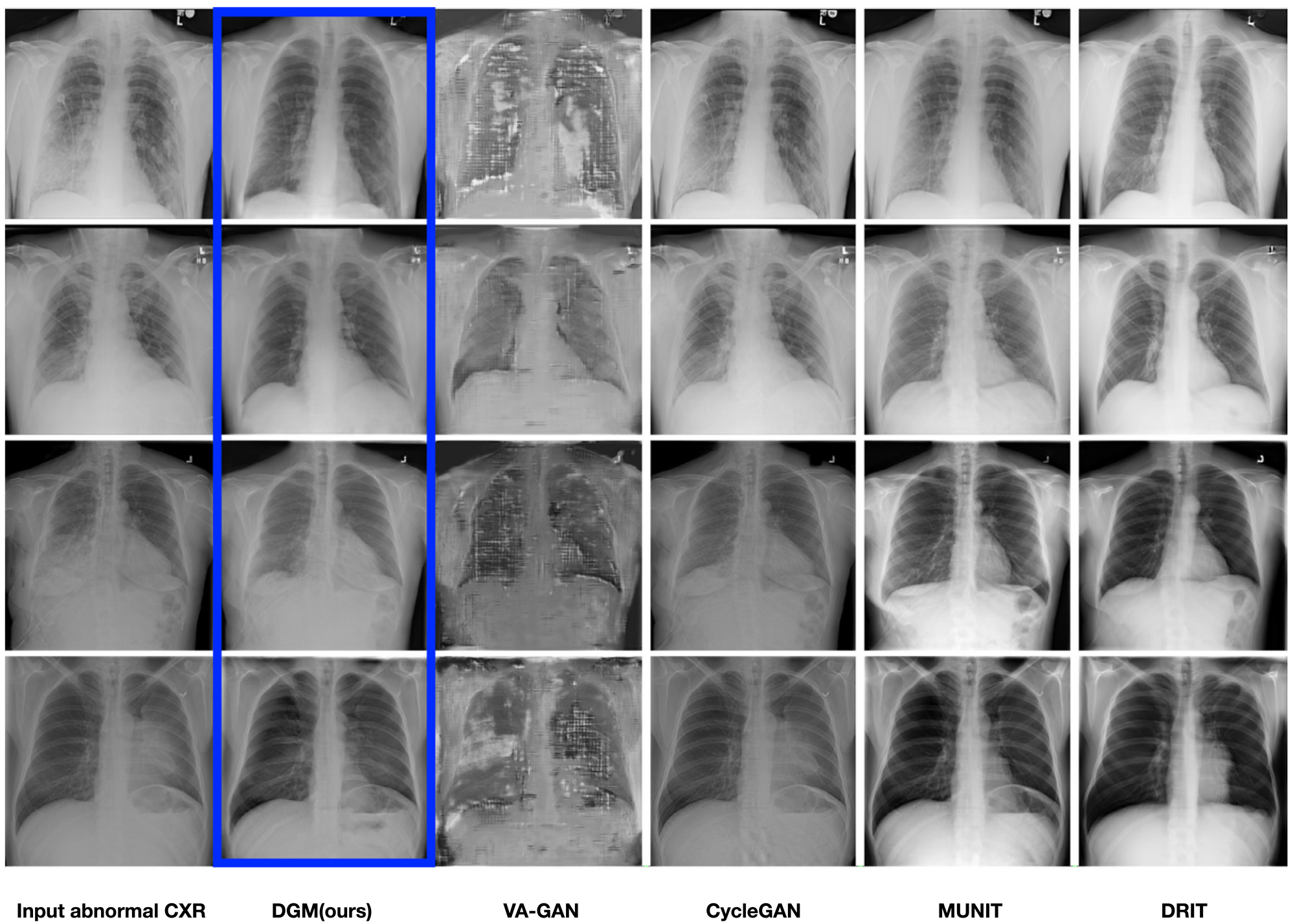
(그림 5)는 residue map과는 반대로 입력 영상이 normal/abnormal이 들어와도 무조건 normal로 생성해주는 Deep Generative Model과 다른 모델들의 비교 결과를 보여주고 있습니다.
마찬가지로 이것도 당연히(?) DGM이 가장 좋은 결과를 보여주고 있는 것을 확인할 수 있습니다.
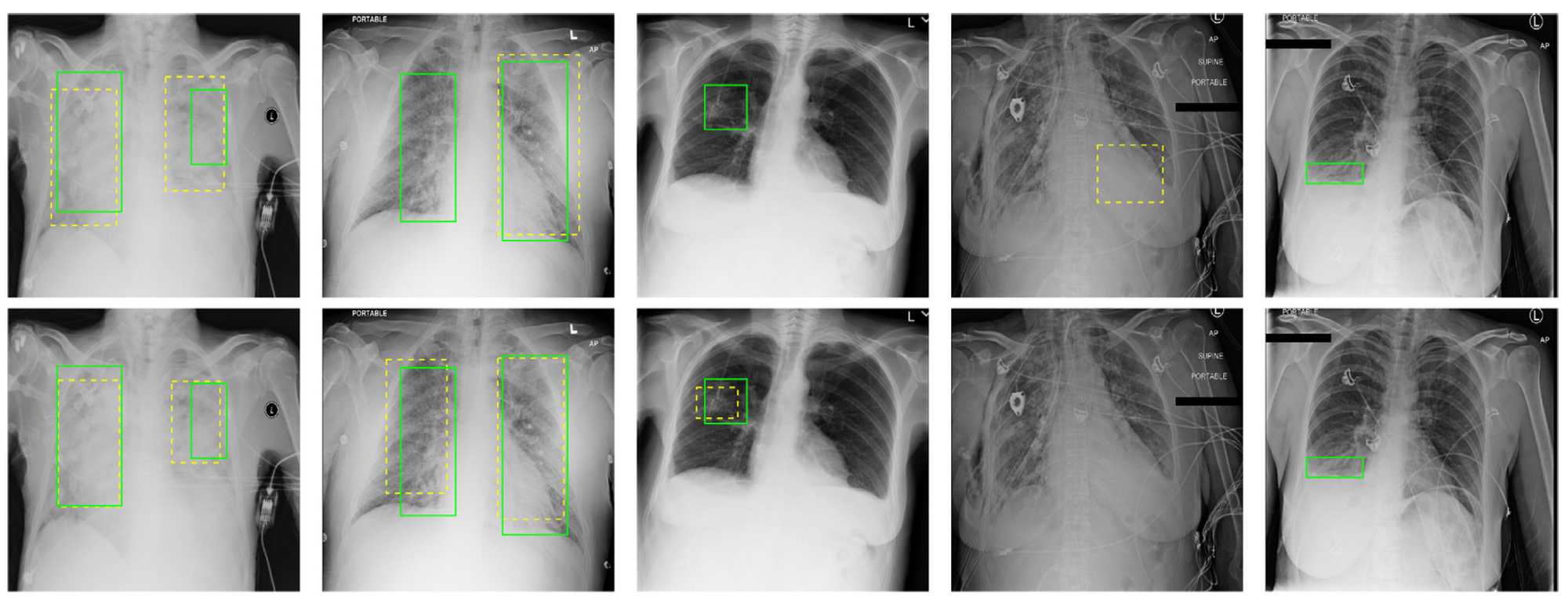
그래서 최종적으로 (그림 7)와 같이 ground trutn(green box)와 prediction results(yellow box)를 비교해보면 잘 찾는다는 것을 확인할 수 있습니다.
Discussion
포스팅한 논문은 Anomaly Detection이지만 F-AnoGAN과 AnoGAN과는 다르게 query image를 normal CXR로 생성해주는 model과 residue map을 구해주는 모델 만들도록 학습을 해야하니 학습 데이터로는 normal/abnormal 데이터가 다 존재햐아 하는 Supervised Learning으로 접근을 하여 Anomaly Detection하는 논문입니다.
본 논문에서는 Unsupervised Learning으로 접근한 방법의 논문들과 비교를 한 흔적은 안보이는데 한번 비교를 하는 방법도 좋은 방법인거 같다는 생각이 들게 하는 논문이였습니다.
